Atrakcyjny kandydat
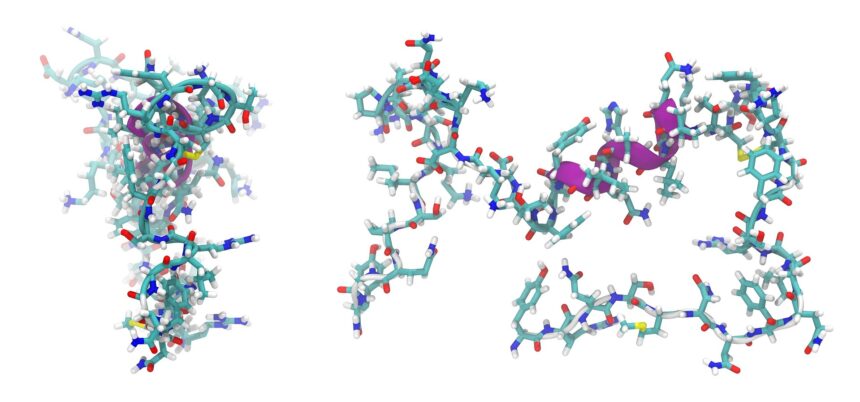
Peptydy to krótkie fragmenty białek, zbudowane z kilkudziesięciu aminokwasów połączonych ze sobą w łańcuch. Docenił je przemysł kosmetyczny i wykorzystuje np. w produkcji kremów, a w farmacji są jednymi z najważniejszych cząsteczek terapeutycznych. Wszyscy znają insulinę, czyli naturalny peptyd regulujący poziom cukru we krwi, który od lat pozostaje jednym z najlepiej sprzedających się leków na cukrzycę. Semaglutyd – znany z leku Ozempic – to peptydowy analog hormonu GLP-1. Zawiera 31 aminokwasów i został sztucznie zmodyfikowany, by dłużej działać w organizmie. Mimo modyfikacji, nadal jest peptydem i stanowi przykład, jak inżynieria białek może znacząco poprawić skuteczność i wygodę terapii.
Atrakcyjność peptydów zwiększa dodatkowo ich wysokie bezpieczeństwo i selektywność – potrafią one celować w konkretne białka związane z chorobą. Mogą być pozyskiwane z roślin lub wytwarzane syntetycznie. Aby działać skutecznie, muszą idealnie dopasować się do „molekularnego zamka”, czyli białkowego receptora. Zaprojektowanie takich peptydów wymaga jednak dokładnej znajomości ich budowy — zarówno składu aminokwasowego, jak i przestrzennej struktury.
Od cząsteczki do leku
Zanim jakakolwiek cząsteczka trafi do farmakologii, musi przejść długą i kosztowną drogę. Wszystko zaczyna się od etapu odkrywania leku – poszukiwania związków, które mogą działać na konkretny cel biologiczny, np. białko związane z chorobą. Następnie badacze sprawdzają, czy dana cząsteczka rzeczywiście wiąże się z tym celem i czy robi to skutecznie. Potem następuje optymalizacja struktury – modyfikacje, które poprawiają działanie, trwałość lub bezpieczeństwo substancji. Kolejne etapy to testy laboratoryjne, badania na zwierzętach i – jeśli wyniki są obiecujące – kosztowne i czasochłonne badania kliniczne na ludziach. Właśnie na tych pierwszych etapach sztuczna inteligencja może zaoszczędzić naukowcom miesięcy, a nawet lat pracy.
Molekularny ślusarz
Peptydy stosowane jako leki zazwyczaj składają się z 5 do 50 aminokwasów. Ponieważ każdą pozycję w łańcuchu może zająć jeden z 20 naturalnych aminokwasów, to liczba możliwych sekwencji nawet w najkrótszym peptydzie wynosi 205, czyli ponad 3 miliony. Dla „przeciętnego” peptydu terapeutycznego składającego się z 20 reszt aminokwasowych, ta liczba rośnie do 2020 możliwych sekwencji – a to ponad 10²⁶ wariantów, czyli milionkrotnie więcej niż ziaren piasku na wszystkich plażach Ziemi! Wyobraźmy sobie ślusarza, który musi dopasować klucz do zamka i ma tyle kluczy do sprawdzenia…
Meta AI – dział badawczy Facebooka – opracował model ESMFold, który przewiduje strukturę białek na podstawie „językowego zrozumienia” ich sekwencji. Choć nie został zaprojektowany do analizy interakcji z peptydami, jego możliwości w tym zakresie zbadał zespół prof. Sebastiana Kmiecika z Wydziału Chemii i Centrum Nauk Biologiczno-Chemicznych Uniwersytetu Warszawskiego, we współpracy z badaczami z Linköping University w Szwecji. Badania przeprowadził doktorant Mateusz Zalewski.
– Okazało się, że radzi sobie z tym zaskakująco dobrze – w wielu przypadkach niemal tak skutecznie jak wyspecjalizowane narzędzia, takie jak AlphaFold, za które w ubiegłym roku przyznano Nagrodę Nobla w dziedzinie chemii. Dzięki zastosowaniu prostych zabiegów obliczeniowych, takich jak np. częściowe ukrywanie fragmentów sekwencji, trafność przewidywanych interakcji przekroczyła 28%. To imponujący wynik jak na model, który nie był trenowany na tego typu danych – wyjaśnia prof. Kmiecik.
Czas, cena, bezpieczeństwo
W projektowaniu leków liczy się, oprócz bezpieczeństwa i ceny wytworzenia, czas. Szczególnie, gdy do sprawdzenia są tysiące potencjalnych kandydatów.
– ESMFold działa błyskawicznie – pojedyncze przewidywanie trwa zaledwie minutę. To czyni go szczególnie atrakcyjnym w projektowaniu leków peptydowych, gdzie trzeba testować setki lub tysiące kandydatów w krótkim czasie – mówi naukowiec.
Dzięki badaniom prowadzonym w CNBCh i na Wydziale Chemii UW jesteśmy o krok bliżej do szybszego i tańszego projektowania leków, które precyzyjnie trafiają w biologiczne cele. Przeprowadzone eksperymenty pokazują, że modele oparte na analizie języka białek mogą być przyszłością biotechnologii.
Link do artykułu: https://pubs.acs.org/doi/10.1021/acs.jctc.4c01585